グラフ作成に必要なパッケージをインストールします。
- phyloseq ―― 構成要素のデータを組み立てるために必要
- ggplot2 ―― グラフ作成に必要
# phyloseqとggplot2をインストールする。
installed.packages("phyloseq")
installed.packages("ggplot2")
#パッケージの呼び出し
library("phyloseq")
library("ggplot2")ggplot2のテーマを指定する。
theme_set() を用いてggplot2 のテーマを指定することで、好みのデザインのグラフを作ることができます。 theme_gray()やtheme_bw()、theme_dark()、theme_light()、theme_linedraw()、theme_minimal()などがあります。
今回は、theme_set(theme_bw())にしてみます。
theme_set(theme_minimal())今回は練習で、仮のOTUデータセットを作成します。
otumat = matrix(sample(1:100, 100, replace = TRUE), nrow = 10, ncol = 10)
outmatというオブジェクトに、1から100までの数字からサンプリングした数字の、縦10行×横10行のデータセットを作成する。
- matrix() ―― 行列の作成
- sample() ―― ランダムにサンプリング(標本を抽出)する
otumat = matrix(sample(1:100, 100, replace = TRUE), nrow = 10, ncol = 10)
otumat
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
[1,] 83 81 50 45 31 84 65 21 60 70
[2,] 61 31 3 82 79 5 79 39 99 83
[3,] 27 40 93 48 27 55 33 73 17 73
[4,] 83 39 40 35 81 85 12 60 52 60
[5,] 51 65 64 32 56 42 32 66 51 45
[6,] 41 96 91 79 33 26 85 78 42 66
[7,] 6 6 62 20 19 32 55 72 100 54
[8,] 14 59 8 80 35 61 86 25 84 88
[9,] 3 5 9 48 77 62 16 22 16 55
[10,] 74 13 27 44 46 21 7 41 25 20 ランダムサンプリングなので、実行ごとに数字が変わります。
otumat = matrix(sample(1:100, 100, replace = TRUE), nrow = 10, ncol = 10)
otumat
[,1] [,2] [,3] [,4] [,5] [,6] [,7] [,8] [,9] [,10]
[1,] 57 45 84 97 5 65 7 62 87 99
[2,] 60 73 40 48 14 3 25 18 46 51
[3,] 71 96 69 29 45 42 86 93 71 43
[4,] 2 38 8 73 37 63 93 51 94 66
[5,] 59 69 14 5 45 8 74 2 45 21
[6,] 85 91 83 1 41 54 12 75 79 35
[7,] 77 64 97 62 12 69 61 74 57 35
[8,] 58 100 38 86 16 84 37 45 39 68
[9,] 54 46 28 90 46 33 99 23 73 67
[10,] 7 99 68 64 14 77 1 42 78 17 行と列に名前を付けます。
rownames(otumat) <- paste0("OTU", 1:nrow(otumat)) #列に名前を付ける
colnames(otumat) <- paste0("Sample", 1:ncol(otumat)) #行に名前を付ける
otumat
Sample1 Sample2 Sample3 Sample4 Sample5 Sample6 Sample7 Sample8 Sample9 Sample10
OTU1 57 45 84 97 5 65 7 62 87 99
OTU2 60 73 40 48 14 3 25 18 46 51
OTU3 71 96 69 29 45 42 86 93 71 43
OTU4 2 38 8 73 37 63 93 51 94 66
OTU5 59 69 14 5 45 8 74 2 45 21
OTU6 85 91 83 1 41 54 12 75 79 35
OTU7 77 64 97 62 12 69 61 74 57 35
OTU8 58 100 38 86 16 84 37 45 39 68
OTU9 54 46 28 90 46 33 99 23 73 67
OTU10 7 99 68 64 14 77 1 42 78 17 paste0() ―― ()の中の要素に文字列を加えて、つなげる。
さっきまで1,2,3,4…だった縦の列に、”OTU”の文字列が加わりました。
行の列には、”Sample”の文字列が加わりました。
仮の分類表のデータも作成します。
taxmat = matrix(sample(letters, 70, replace = TRUE), nrow = nrow(otumat), ncol = 7)
rownames(taxmat) <- rownames(otumat)
colnames(taxmat) <- c("Domain", "Phylum", "Class", "Order", "Family", "Genus", "Species")
taxmat
Domain Phylum Class Order Family Genus Species OTU1 "u" "o" "n" "e" "q" "w" "a"
OTU2 "j" "z" "h" "b" "d" "w" "n"
OTU3 "a" "d" "v" "m" "n" "a" "c"
OTU4 "y" "h" "i" "f" "z" "s" "v"
OTU5 "i" "n" "l" "t" "a" "o" "r"
OTU6 "l" "q" "c" "g" "g" "q" "i"
OTU7 "c" "s" "g" "v" "z" "w" "h"
OTU8 "u" "f" "s" "b" "n" "q" "g"
OTU9 "j" "u" "y" "w" "d" "h" "o"
OTU10 "i" "q" "d" "e" "q" "r" "d"otu_table ―― OTUの豊富さのオブジェクトの構築
tax_table ―― 分類名の表を作成する
library("phyloseq")
OTU = otu_table(otumat, taxa_are_rows = TRUE)
TAX = tax_table(taxmat)
OTU
OTU Table: [10 taxa and 10 samples] #10分類と10サンプル
taxa are rows
Sample1 Sample2 Sample3 Sample4 Sample5 Sample6 Sample7 Sample8 Sample9 Sample10
OTU1 57 45 84 97 5 65 7 62 87 99
OTU2 60 73 40 48 14 3 25 18 46 51
OTU3 71 96 69 29 45 42 86 93 71 43
OTU4 2 38 8 73 37 63 93 51 94 66
OTU5 59 69 14 5 45 8 74 2 45 21
OTU6 85 91 83 1 41 54 12 75 79 35
OTU7 77 64 97 62 12 69 61 74 57 35
OTU8 58 100 38 86 16 84 37 45 39 68
OTU9 54 46 28 90 46 33 99 23 73 67
OTU10 7 99 68 64 14 77 1 42 78 17 physeq = phyloseq(OTU, TAX)
physeqphyloseq() ―― 構築メソッドの一つ。構成データから得られる実験レベルオブジェクトを作成できる。
グラフを出力します。
plot_bar(physeq, fill = "Family")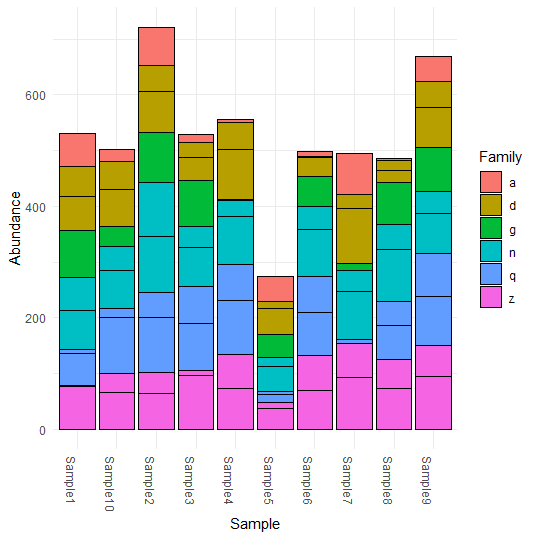
参考:http://joey711.github.io/phyloseq/import-data

こんにちは。
大学で土壌中の真菌について研究しているものです
phyloseqとggplotsをCRANからインストールしたときのRのバージョンを教えていただきたいです。
こんにちは。ご質問ありがとうございます。
ggplot2は3.3.6、phyloseqは1.38.1だったかと思います。
ご返信ありがとうございます。
質問が分かりにくく申し訳ありません。
パッケージをインストールした時の’R自体’のバージョンです。
お手数をお掛けしますが、よろしくお願い致します。
大変失礼しました。4.1.1だったと思うのですが、記憶が曖昧です。
すみません…。
ご返信ありがとうございます!
いえいえ!研究頑張ってください。